新型コロナウイルス寄稿論文(『生物の科学 遺伝』Special)
|
豚で起きていたコロナウイルスに近縁な |
|
ウイルスでのパンデミック感染の紹介 |

花田 耕介
Hanada Kousuke
九州工業大学大学院情報工学研究院准教授
|
花田 耕介 |
|
|
Hanada Kousuke |
|
|
九州工業大学大学院情報工学研究院准教授 |
|
|
略歴 |
|
|
2000-2003年,総合研究大学院大学生命科学研究科遺伝学専攻(理学博士)。2003-2004年,国立遺伝学研究所 ポスドク研究員。2004-2005年,米国シカゴ大学 ポスドク研究員。2006-2007年,米国ミシガン州立大学 ポスドク研究員。2007-2012年,理化学研究所 研究員。2012-2017年,九州工業大学・若手フロンティア研究アカデミー・准教授。2017年現在,九州工業大学・大学院情報工学研究院・生命情報工学研究系・准教授。専門分野は,ゲノム解析。理化学研究所・研究奨励賞(2012年3月)「Breakthrough in evolutionary mechanism of duplicate genes based on bioinformatics」,日本進化学会研究奨励賞(2014年8月)「網羅的なゲノム情報を利用した進化ゲノミクス研究」受賞。主な著書に,進化学辞典. 第二章ウイルスと原核生物(DNAウイルス) P.26-29 (共立出版, 2012), バイオディバーシティシリーズ 第4巻 菌類の多様性と系統(地衣類,細菌,ウイルスを含む)ウイルスの多様性と分類 P.369-395(共著, 昇華房, 2005)。 |
|
|
COVID-19は世界中に爆発的な感染拡大(パンデミック)が起きている。これらと同じような感染を豚に引き起こしたウイルスはあまり知られていない。ここでは,豚に30年前にパンデミックを引き起こした豚繁殖・呼吸障害症候群ウイルス(PRRSV: Porcine Reproductive and Respiratory Syndrome Virus)の進化研究を紹介する。PRRSVは,パンデミックを引き起こすことで,異常な複製回数を引き起こし,進化速度が加速化し,多様なウイルスが出現していた。このまま,COVID-19のパンデミックが続き,パンデミックが起きた国で隔離が行われるとPRRSVと同様にCOVID-19にも多様性が生まれることが予想される。 |
WHOの定義では,エマージング感染症(新興感染症)は,「新たにヒトや動物の集団の中に出現した感染症」とされている。このようなエマージング感染症が引き起こすウイルス感染症の中でも,マールブルク病,ラッサ熱,エボラ出血熱のように致死率が高いものは,感染した患者の隔離が容易になるので,爆発的な感染拡大(パンデミック)は起きにくい。
今回,世界中で蔓延しているコロナウイルス(Coronavirus Disease 2019:COVID-19)は,これらのエマージング感染症に比べて致死率が低く,不顕性感染でウイルスを保有し続ける患者も多いため,感染した患者の隔離が困難である。そのため,パンデミックに繋がる可能性が高かった。残念ながら,COVID-19の感染は,世界中に広がり,2020年4月5日時点では,感染者が110万人を超え,1日1万人ずつ増加している過去に例をほとんど見ない感染症になった1)。
著者は,COVID-19が世界中に感染拡大を引き起こす様子を見ながら,筆者が約15年前に行った博士課程の研究「30年前に豚でパンデミックを引き起こしたコロナウイルスに近縁なウイルス」との類似点があると考えた。そこで,自分の研究を簡単に紹介した後に,コロナウイルスがパンデミックによってどのような進化がおきるかが予想されるか議論していきたいと考えている。
豚繁殖・呼吸障害症候群ウイルス(PRRSV: Porcine Reproductive and Respiratory Syndrome Virus)は,アルテリウイルス科に分類されるが,コロナウイルスと同じ,ニドウイルス目に属し,ゲノムサイズは異なるものの,ゲノム構造は非常に類している(図1)2) 。
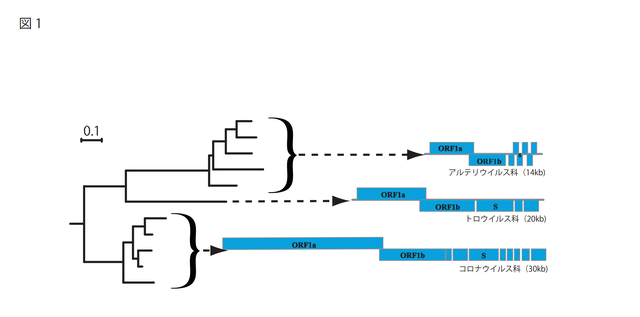
図1.ニドウイルス目の系統樹
青の四角は,一つのOpen Reading Frameを意味する。ORF1aとORF1bの保存領域を李利用して,アミノ酸配列を用いて近隣結合法でニドウイルス目の分子系統樹を構築した。括弧の中はゲノムサイズを示す。
このウイルスは,1980年の中頃に北米で新興感染症として認識されてから約5年間で世界中に拡散し,二つの型(北米型および欧州型)が存在していることが明らかとなっている3)4)。ウイルスは全ての日齢の豚に感染し,主に肺炎を種とする呼吸器症状と流産を引き起こし,体細胞のいたるところでウイルスが増殖できる。その結果,鼻汁,唾液,尿,糞便,精液などの体液にウイルスが排泄され,接触,飛沫および交配による水平感染や垂直感染,そして隣接する農場等では風による伝播が成立する。このように,PRRSVは感染能力が極めて高いウイルスである。1992年にこれらの感染症を引き起こすと考えられる原因ウイルスが分離されると,ウイルスには北米で分離された株と欧州で分離された株にはアミノ酸配列の類似性が60%しかないことが明らかとなった5)6)。ここまでの現象を整理すると,1980年の中頃に北米で見出されたものから,10年以内で,40%も異なる北米型と欧州型の二つに分岐したと考えられた。しかし,今まで報告されているウイルスの進化速度で考えても,10年間でアミノ酸配列の類似性が40%も異なるものに変化したとはとは考えにくい。
そこで,著者らは,1990年以降に北米で大量に分離された株と欧米株のDNA配列を比較し,北米株の分離年代から最近になるにしたがって,一定の割合で欧州型から北米型にDNA配列が異なっていることを発見した7)。DNA配列が異なる速さ(進化速度)は,5.6 - 9.8×10-2(塩基置換数/塩基/年)と1年あたり,100塩基あたり平均して5.6から9.8個の塩基が変化していることが明らかとなり,その進化速度の速さは今まで推定されたどのRNAウイルスの進化速度よりも早いことを明らかとした(図2)。
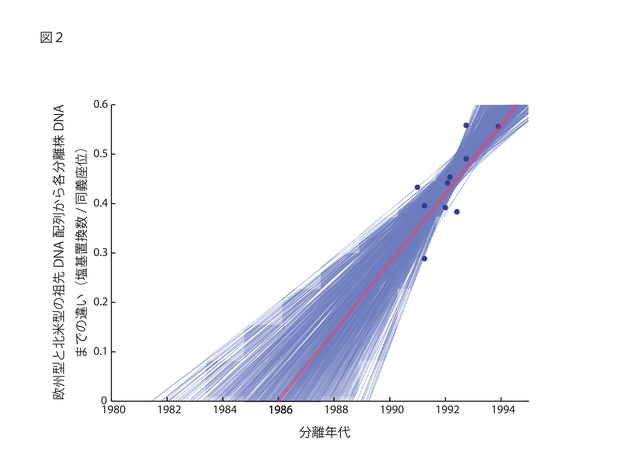
図2.PRRSVの欧州型と北米型が分岐年代の推定
Hanada K, et al. 2005の論文で使用されている図を改変した。青丸は北米型の分離株を示す。X軸とY軸は,それぞれ分離年代と欧州型と北米型の祖先型からの進化距離を示す。進化距離はアミノ酸が変化しない同義座位に着目し同義座位の塩基置換数で示す。最小二乗法により欧州型と北米型の分岐年代を推定した(赤線)。さらに、その分岐年代のばらつきを、ブートストラップ法で推定した(青線)。
そして,この速さで進化すると,北米型と欧州型が分岐したのは1984―1988年であることが推測できた。つまり,現在みられる北米型と欧州型は,1980年代の中頃に北米の豚で新興感染症として認識されたものから,大きく進化し,60%しか類似性が見られないウイルスに変化したことを意味している。
ここで,PRRSVは,なぜ,このような速さで進化できたのかという疑問がでる。そこで,著者は,PRRSVだけでなく,約50種類のRNAウイルスの進化速度を推定し,どのようなウイルスで進化速度が速く,どのようなウイルスで進化速度が遅いのかを検討した。その結果,感染するチャンスが多い感染様式を持つ(飛沫感染等)ウイルスの進化速度がはやく,感染するチャンスが少ない感染様式持つ(蚊媒介,血液感染)ウイルスの進化速度が遅いことを発見した(図3)8)。
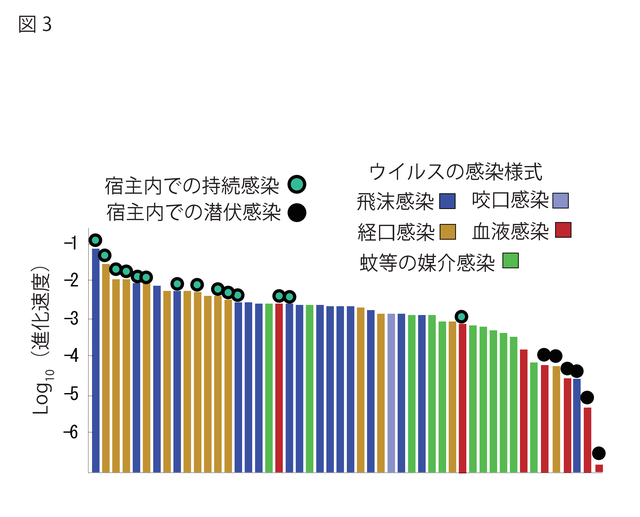
図3.ウイルス進化速度と感染様式の関係
Hanada K, et al. 2004の論文で使用されている図を改変した。49種類のRNAウイルスの進化速度を推定し上位から並べた。宿主内で持続感染するウイルスは継続的に宿主内で複製するウイルスであり,宿主内で潜伏感染ウイルスは宿主内でウイルス複製をしないウイルスである。持続感染するウイルスの方が潜伏感染するウイルスよりも進化速度が速くなる傾向があった。感染が成立しやすい感染様式(飛沫感染,経口感染)は,感染が成立しにくい感染様式(血液感染,蚊等の媒介感染)に比べ,進化速度が速くなる傾向があった。
これは,非常に理解できる現象である。なぜならば,進化速度は,1年あたりに起きるDNA塩基の変化率である。これは,「一回の複製で生じる複製エラー率」,と,「複製の回数」の積になる。一回の複製エラー頻度は,RNAウイルス間で大きくは変わらないため,1年あたりに起きる複製の回数が,進化速度を決定する主要因であることが示唆された。そう考えると,感染するチャンスが多いウイルスは,複製回数が大きいことが期待できる。その結果,感染能力が高いウイルスは非常に高速度で進化している現象が見出されたのである。つまり,豚に感染するPRRSVは,一端豚舎に侵入すると,大量の豚に感染する。そこで,数多くの複製を引き起こす。さらに,感染能力が非常に高く,数キロ離れた豚舎所にも侵入し感染するPRRSVは,短期間に大量の豚に感染し,異常な量の複製を成し遂げたと予測できる。その結果,今まで報告されたどのウイルスよりも進化速度が高くなったと考えられた。
これらのことを,現在,蔓延しているCOVID-19に当てはめてみると,COVID-19も異常なスピードで進化をし続けていると容易に予測できる。COVID-19は,非常に高い感染能力を持つウイルスであることは明らかである。今後,国ごとに隔離をすることによって,国ごとに異なる型が生まれていることも予想できる。つまり,今後は,さまざまな地域で感染し続けているCOVID-19の多様性を明らかにし,どのようなCOVID-19においても効果が高いワクチン開発あるいは治療薬の開発を行う必要があると考えられる。
(2020/4/7)
文献
1. WHO. 取得日 2020年4月6日 <https: //www.who.int/emergencies/diseases/novel-coronavirus-2019>
2. Cavanagh, D. 1997. Nidovirales: a new order comprising Coronaviridae and Arteriviridae. Arch. Virol. 142:629–633.
3. Keffaber, K. K. 1989. Reproductive failure of unknown etiology. Am. Assoc. Swine Pract. Newslett. 1–9.
4. Albina, E. 1997. Epidemiology of porcine reproductive and respiratory syndrome (PRRS): an overview. Vet. Microbiol. 55:309–316.
5. Wensvoort, G., E. P. de Kluyver, E. A. Luijtze, A. den Besten, L. Harris, J. E. Collins, W. T. Christianson, and D. Chladek. 1992. Antigenic comparison of Lelystad virus and swine infertility and respiratory syndrome (SIRS) virus. J. Vet. Diagn. Invest. 4:134–138.
6. Murtaugh, M. P., K. S. Faaberg, J. Laber, M. Elam, and V. Kapur. 1998. Genetic variation in the PRRS virus. Adv. Exp. Med. Biol. 440:787–794.
7. Hanada K1, Suzuki Y, Nakane T, Hirose O, Gojobori T. 2005. The origin and evolution of porcine reproductive and respiratory syndrome viruses. Mol Biol Evol. 22(4):1024-1031
8. Hanada, K., Y. Suzuki, and T. Gojobori. 2004. A large variation in the rates of synonymous substitution for RNA viruses and its relationship to a diversity of viral infection and transmission modes. Mol. Biol. Evol. 21:1074–1080.





